DNA 甲基化测序(Bisulfite-Seq)的比对和检测工具Bismark原理浅析
好久没更新了,最近在弄DNA甲基化测序的东西,接触到了Bismark这个软件,由于对甲基化测序数据的比对有点疑惑,同时文献不长,所以简单看了下,在此做个记录。
一、前言
将DNA使用重亚硫酸盐处理,使得DNA链中的碱基非甲基化C转为U,而后在PCR流程中转化为碱基T,而甲基化的碱基C不受影响(更详细的建议自行查找下材料、文献学习)。亚硫酸盐文库有两种不同的类型:定向测序,主要包括正向序列及其互补序列的转换序列;而第二种是包括了4种情况的序列;
二、 算法浅析
对于比对的算法,由于我们的序列经过C-T,G-A转换(相当于互补链的C-T转换),所以直接用常规的方法进行比对肯定是会有问题的,所以作者将序列进行了C-T,G-A转换,同时将参考序列也进行了该处理;而后将处理后的序列使用bowtie比对到转换后的参考序列上
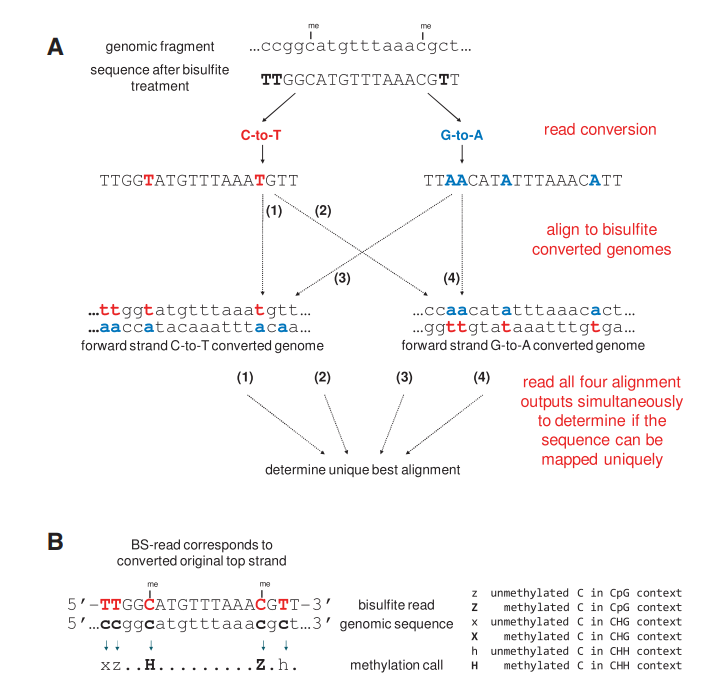
图一:比对示意图
同时,bismark中的cpg位点有三种情况:CpG、CHG、CHH(其中H表示A、T或C),用z、x、h分别表示这3种情况,小写表示未甲基化,大写表示甲基化。
三、参考
1. Krueger, Felix, and Simon R Andrews. “Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications.” Bioinformatics (Oxford, England) vol. 27,11 (2011): 1571-2. doi:10.1093/bioinformatics/btr167
版权声明:本文转载请注明出处!



最新评论:
发表评论
电子邮件地址不会被公开。 必填项已用*标注