引物特异性验证
作为生物dog,相信分子生物学实验做的不少(当然,因为专业原因,我虽然做过,但是很少),而这个往往离不开引物设计,在引物设计完成后,我们如何验证引物的特异性,防止扩增的时候引入杂带呢?下面我将介绍两种方法进行校验,希望能对你有所帮助!
一、primer-blast
primer-blast是NCBI平台下的一个引物设计软件,其使用Primer3 设计 PCR 引物,同时使用blast和全局比对算法在用户选择的数据库中进行检索,来避免非特异性的引物。所以如果你是用primer-blast进行引物设计的话,可根据其结果进行引物特异性的判断,或者你也可以将你从其他地方设计的引物输入到primer-blast程序进行判断,这里将介绍后一种方法。
首先打开primer-blast官网 https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi ,在对应的位置输入相应的引物序列,PCR Template留空即可,其他参数默认。
这里我们从网上随意找一对引物
F: CAGCCCCCTTCGCCCCGAGAGGGCCCTCTGCCT
R: ACCCTAGAGTCTCATTGGGCAGGGCC
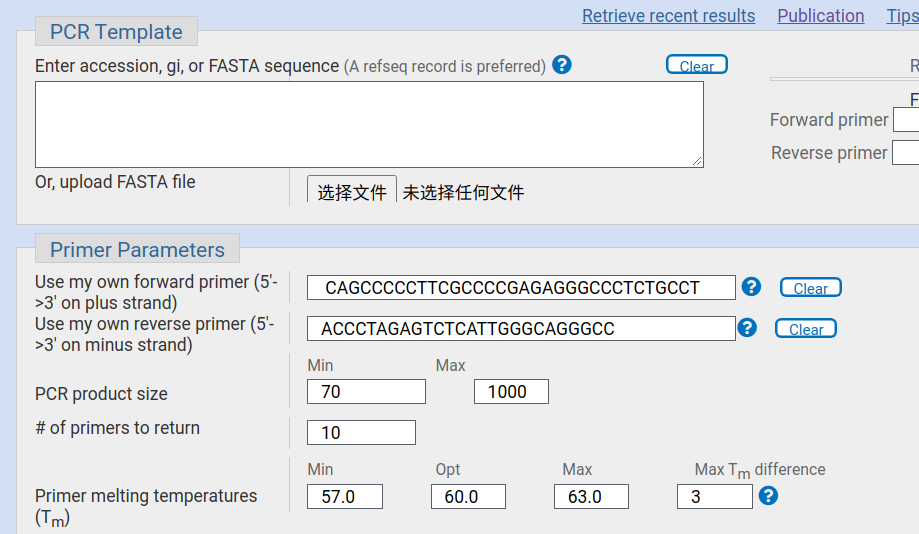
图1-1:输入引物信息
输入完引物序列后,在下方选择需要比对到的数据库,以及对应的物种
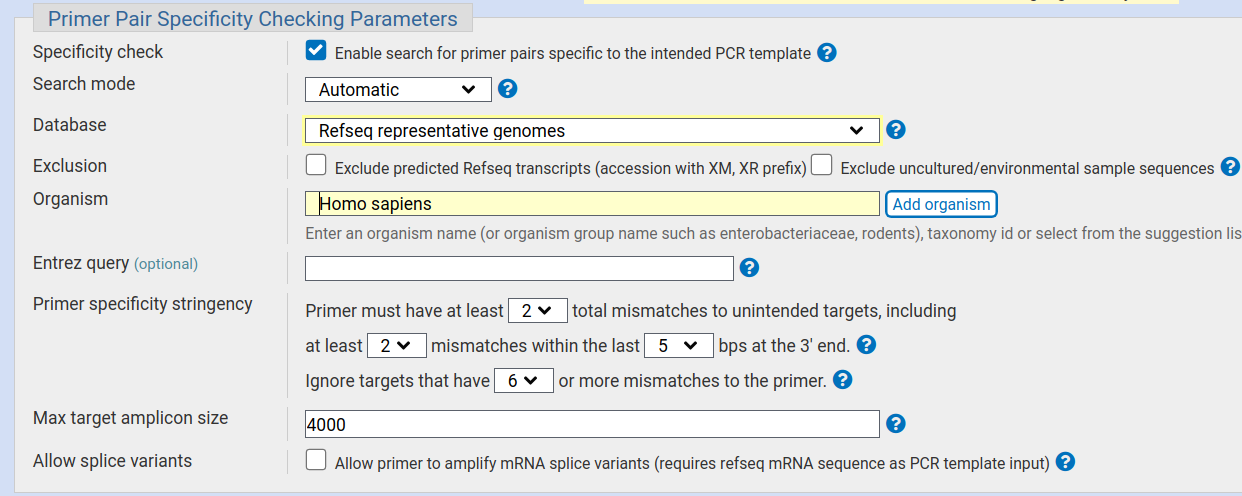
图1-2:选择数据库及物种
上诉全部输入完成后,点击Get Primers 运行程序即可。等一会结果就会出来了,像下诉这种结果我们可以看到只有一条记录,这种情况下特异性就很好了,如果有多个记录的话,看是否能通过长度等条件筛选,如果不能的话,建议重新设计过吧。
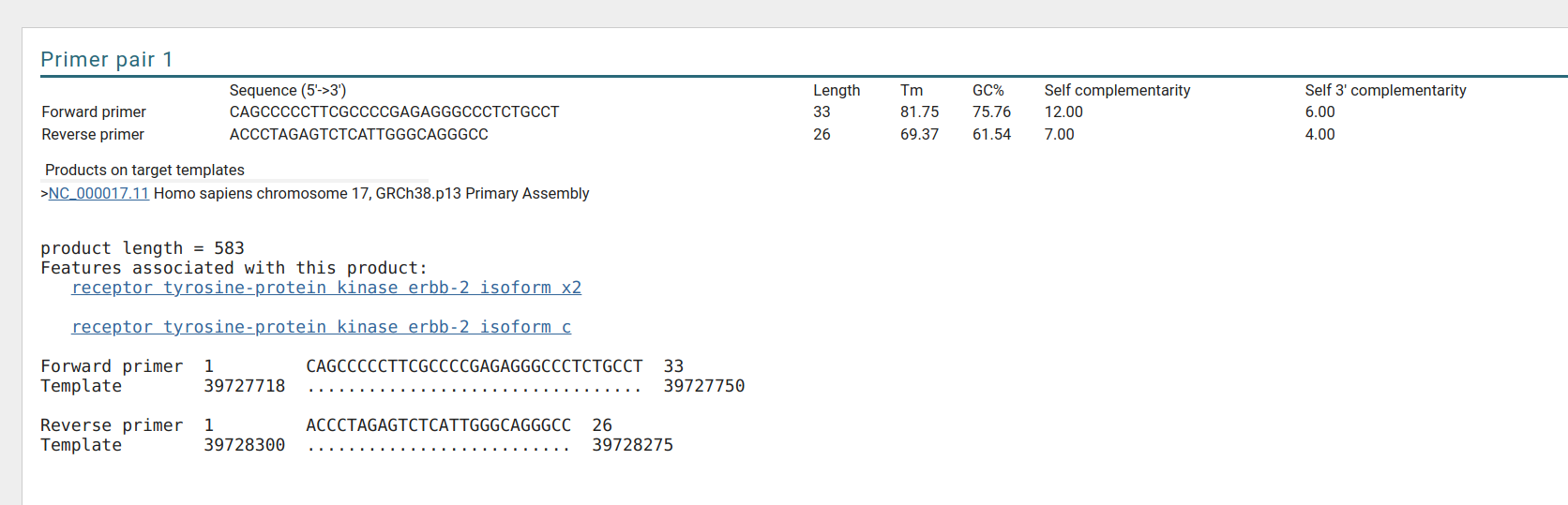
图1-3:结果
二、blast
除了用primer-blast来进行引物特异性验证外,我们还可以直接使用blast来进行。
首先打开blast官网 https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome ,将前置引物和后置引物按顺序连接起来,在中间输入几个n(代表未知碱基,或者你也可以分别对前置引物和后置引物进行blast),而后在下方选择对应的数据库和物种。
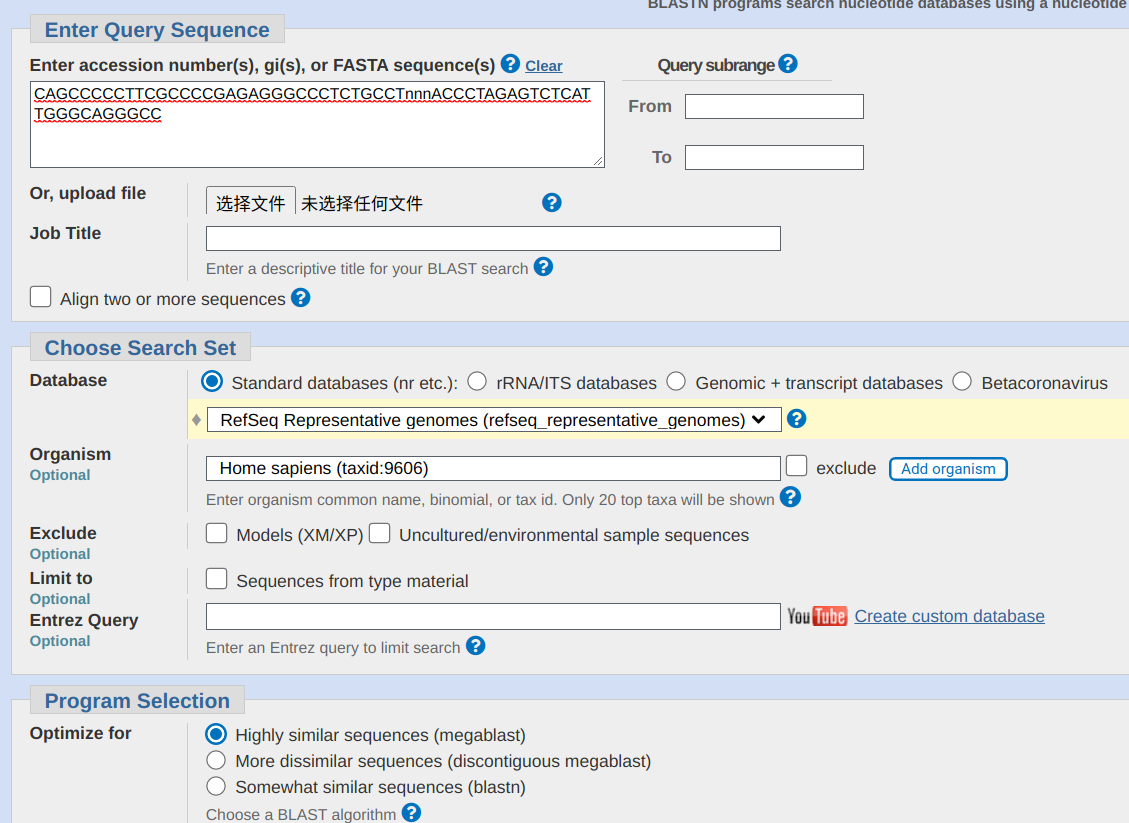
图2-1:引物输入
其次,比较重要的是,需要在算法参数中,将word size更改为比前置引物以及后置引物长度都要小的值,否则对结果会有影响,其他参数可默认。
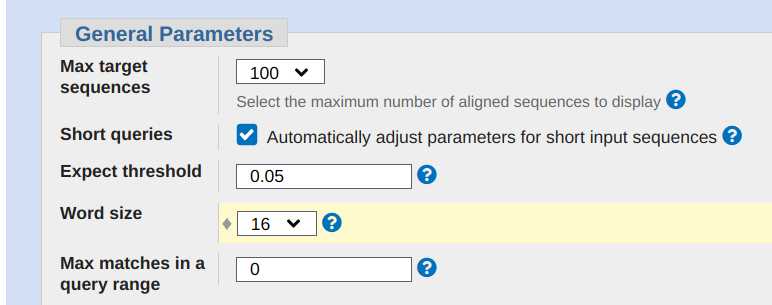
图2-2:算法参数设置
设置完后点击blast即可。
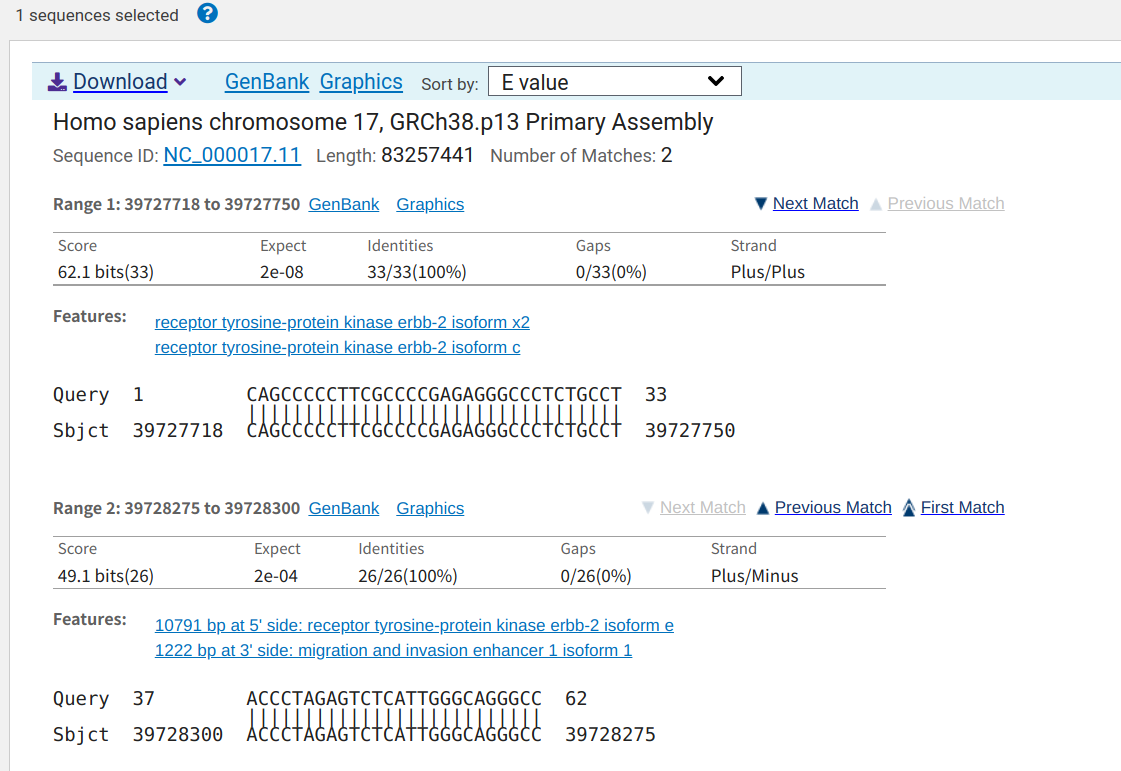
图2-3:结果
我们从上诉结果中可以看到,前置引物和后置引物均比对上了,对应到参考序列的位置和primer-blast的结果是一致的。
三、总结
使用primer-blast或者blast进行引物特异性识别都是可行的,使用primer-blast相对来说比较方便,但是对于批量引物验证比较麻烦,因为其没有本地版,而使用blast则可以使用本地版,编写程序来进行批量处理。当然,由于个人学识有限,同时对于实验接触的少,难免有疏漏的地方,欢迎大家指正!共同学习,共同进步!




最新评论:
发表评论
电子邮件地址不会被公开。 必填项已用*标注