Blast本地安装使用(windows平台)
一、简介
BLAST(Basic Local Alignment Search Tool)是一种广泛使用的生物信息学工具,用于在生物数据库中搜索相似序列。其基于局部比对算法,可以将输入的核酸或蛋白质序列与数据库中的已知序列进行比对,获得序列相似度等信息,从而判断序列的来源或进化关系。在我们日常的科研活动中,我们或多或少都接触过blast,不用一般都是用的网页端,但是网页端有一定的局限性:
- 上传文件不能太大
- 速度慢
- 不能使用自定义数据库
- 数据安全性(敏感数据在本地比对更安全)
为了解决这个问题,我们需要在本地电脑上安装blast,下面我将介绍下如何进行安装以及其简单使用(linux平台下的安装使用见之前的文章-Linux 安装Blast本地版)
二、Blast安装
2.1 下载
为了安装Blast,首先我们先打开blast的官网(https://blast.ncbi.nlm.nih.gov/blast/Blast.cgi),然后点击Download BLAST,在打开的页面中点击下载链接(https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/),然后下载windows平台下的安装包,同时也可以将安装包对应的md5文件一并下载(md5文件主要为了校验下载文件的完整性,下不下载均可以)


2.2 安装
2.2.1 安装包完整性校验(此步可不做)
文件下载完成后,我们可以先校验下安装包的完整性,其具体步骤如下:
进入到安装包的目录,按住左边Shift的同时,点击鼠标右键,点击打开powershell或者终端,在出现的终端下输入下述指令(具体文件名以你下载的为准)
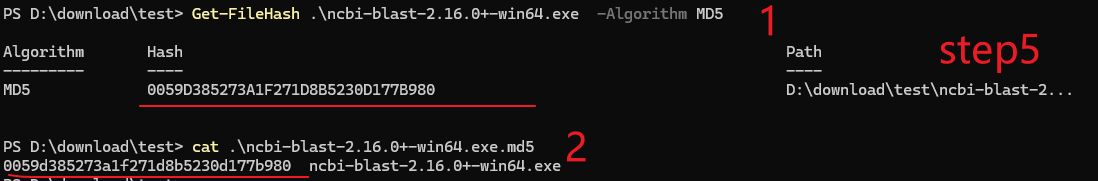
Get-FileHash .\ncbi-blast-2.16.0+-win64.exe -Algorithm MD5
按下回车键后即可以计算出安装包的MD5,再输入下述命令查看下载的MD5文件内容
cat .\ncbi-blast-2.16.0+-win64.exe.md5
若输出的MD5 序列完全一致,则说明下载的安装包是完整的
2.2.1 blast安装
这一步的安装较为简单,直接双击下载的安装包文件,按照提示操作即可完成安装
三、本地blast的简单使用
安装完成后,在安装目录下会有比较多的运行程序,这里我们主要介绍常用的几个,并以其中一个作为例子
|
程序 |
功能简介 |
|---|---|
|
makeblastdb |
将FASTA文件转为blast数据库 |
|
blastn |
核酸序列在核酸库搜索 |
|
blastp |
蛋白序列在蛋白库搜索 |
|
blastx |
核酸序列在蛋白库搜索 |
|
tblastn |
蛋白序列在核酸库搜索 |
|
update_blastdb.pl |
在NCBI中下载BLAST数据库 |
3.1 数据库构建
要使用本地blast服务,必须要下载或者构建本地数据库,这里我介绍3种方法:
- 我们可以在NCBI下载相应物种的数据库(https://ftp.ncbi.nlm.nih.gov/blast/db/),例如下载nr库,需要把对应的nr库全部下载下来。

- 或者也可以使用下面的第一个命令下载对应的数据库(该命令需要预先安装perl环境,终端打开可以用上诉shift+鼠标右键的方法,也可以同时按住win+R键,在弹框中输入cmd,回车即可)
update_blastdb.pl --decompress nr [*] # 下载核酸数据库
update_blastdb.pl --showall [*] # 显示可下载的库
update_blastdb.pl --help # 查看帮助
- 或者我们也可以用makeblastdb命令将fasta文件转为数据库,即自定义数据库。我们可以将nr库序列或者其它需要作为数据库的fasta序列作为输入进行构建数据库,其命令如下:
makeblastdb -in sequences.fasta -input_type fasta -title test_title -out test_out -dbtype nucl
上诉命令中,in后接的是输入的建库序列的路径;input_type是输入序列的文件类型;title是指数据库的标题,out是指数据库的数据路径及名称,后续使用该数据库搜索时需要指定;dbtype是指构建的数据库类型,nucl为核酸库,prot为蛋白库;帮助文档可以输入makeblastdb -help查看,
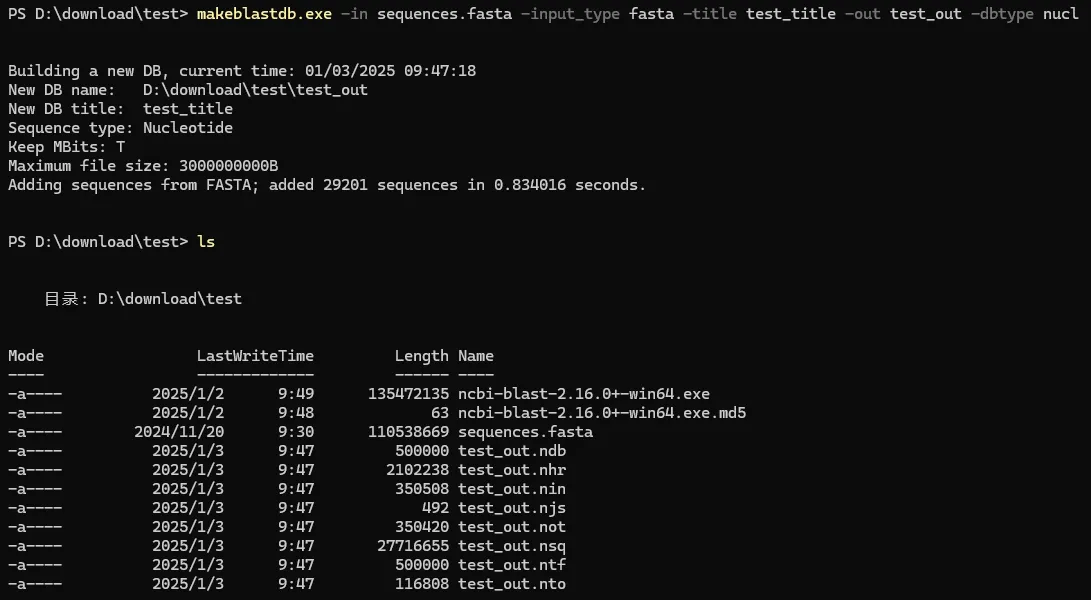
可以看到,当数据库构建成功后,将在目录下生成以test_out为前缀的一系列文件,这些就是构建好的数据库文件。
3.2 本地blast搜索
基于上诉的步骤我们已经得到了一个数据库,现在我们就可以用一些序列来进行测试搜索了,其命令如下所示:
# 以下命令根据需求选择其一即可
blastn -query exam.fa -db test_out -out exam_result.html -html # 输出html格式,可以直接用浏览器打开
blastn -query exam.fa -db test_out -out exam_result.tsv -outfmt 7 # 输出tsv格式
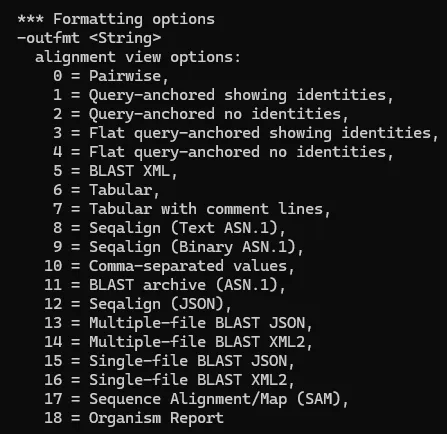
上诉命令中query后接的是需要查询的序列文件,fasta文件格式,可以是单条序列,也可以是多条序列;db是搜索数据库的路径及名字,这里我们选择的是上面创建的数据库test_out;out是输出的文件路径及名字,仅有一个文件名说明输出到当前目录下;html 表示输出html格式,或者也可以指定outfmt参数,从而设定不同的输出格式,例如这里设置了7,说明输出的是TSV格式;blast的输出格式可以通过outfmt参数来进行配置,不同的数字表示不同的格式,见下图所示:
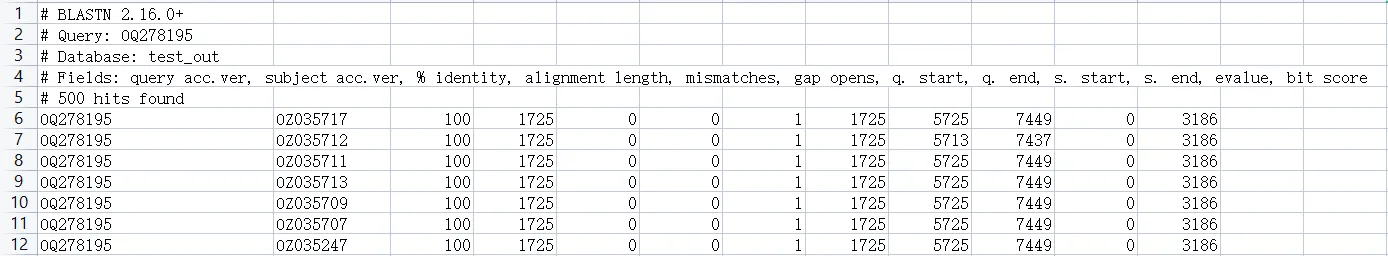
blastn输出的部分结果见图所示(格式7),我们可以看到第4行有每列的列名,其中query acc.ver是查询序列的ID, subject acc.ver是数据库序列ID, % identity表示一致性, alignment length表示比对长度, mismatches表示错配情况, gap opens表示空位情况, q. start是查询序列比对后的起始位置, q. end是查询序列比对后的终止位置, s. start是数据库序列比对后的起始位置, s. end是数据库序列比对后的终止位置, evalue表示期望值,越小越好, bit score表示bit得分,越大越好。
上诉是blastn的一个简单示例,其它的软件,像blastp等也是一样的用法,只不过需要把输入的核苷酸序列换成蛋白质序列,核酸数据库换成蛋白数据库即可。
以上就是blast在windows平台下的安装以及简单使用,如果想要定制输出格式等更复杂的功能,可以通过软件 -help参数查看具体的使用参数!



最新评论:
发表评论
电子邮件地址不会被公开。 必填项已用*标注