基于HMM模型的kegg注释软件KofamKOALA
KEGG数据库是了解生物系统(例如细胞、生物、生态系统)的实用性数据库,是国际最常用的生物信息数据库之一。一般进行kegg注释我们都是通过kegg网页版或者其API进行提交,今天我们介绍一个基于hmm模型进行本地kegg注释的软件KofamKOALA。
KofamKOALA 基于KOfam(KEGG Orthologs (KOs)定制的HMM数据库),通过使用HMMER/HMMSEARCH软件将KO号分配到用户提交的序列上。高于预定义阈值的KO号可信度更高,并且在输出文件中会有*号前缀。同时网页版的KofamKOALA会自动将KO结果发送到KEGG Mapper,以重建通路信息(PATHWAY), pathway modules (MODULE)和hierarchical function classifications (BRITE)。
一、安装
官网:https://www.genome.jp/tools/kofamkoala/
准备:
- Linux
- Ruby >= 2.4
- HMMER >= 3.1
- GNU Parallel
到官网下载KOfam和KofamScan

图1:下载
将下载的kofam数据库解压,确保数据库有profiles目录and ko_list文件,将下载的软件KofamScan解压即可使用
下载安装命令如下:
mkdir -p ~/kofamscan/db
cd ~/kofamscan/db
wget ftp://ftp.genome.jp/pub/db/kofam/ko_list.gz
wget ftp://ftp.genome.jp/pub/db/kofam/profiles.tar.gz
gunzip ko_list.gz
tar xvzf profiles.tar.gz
mkdir -p ~/kofamscan/bin
cd ~/kofamscan/bin
wget ftp://ftp.genome.jp/pub/tools/kofamscan/kofamscan.tar.gz
tar xvzf kofamscan.tar.gz
二、使用
KofamKOALA的运行命令为exec_annotation,运行方式有两种
- 命令行传参
- 使用配置文件
编辑配置文件
cd ~/kofamscan/bin/
cp config-template.yml config.yml
#config.yml示例:
#profile: /path/to/home/kofamscan/db/profiles
#ko_list: /path/to/home/kofamscan/db/ko_list
#hmmsearch: /path/to/home/anaconda3/bin/hmmsearch
#parallel: /path/to/home/anaconda3/bin/parallel
#cpu: 8
使用配置文件运行
exec_annotation -c config.yml
结果示例如下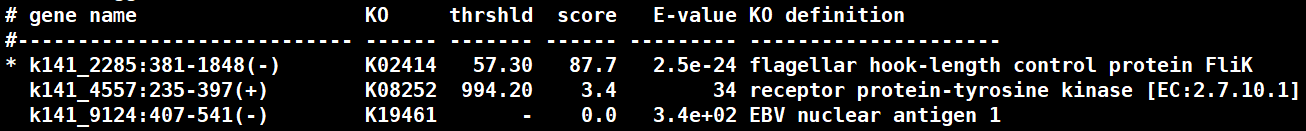
图2:结果
参考:
- Aramaki T., Blanc-Mathieu R., Endo H., Ohkubo K., Kanehisa M., Goto S., Ogata H.
KofamKOALA: KEGG ortholog assignment based on profile HMM and adaptive score threshold.
Bioinformatics. 2019 Nov 19. pii: btz859. doi: 10.1093/bioinformatics/btz859.
版权声明:本文转载请注明出处!



最新评论:
发表评论
电子邮件地址不会被公开。 必填项已用*标注