蛋白互作网络分析(PPI)
蛋白质互作网络分析对了解蛋白质功能、关系有重要意义。一般来说,现在分析蛋白质互作网络使用的是string在线数据库进行分析,而后再使用cytoscape对网络进行下一步的分析或修整。
一、string数据库的使用
打开官网,点击Multiple Proteins ,在输入框中输入差异表达基因名,一行一个,选择好物种后,点击search,而后再选择continue
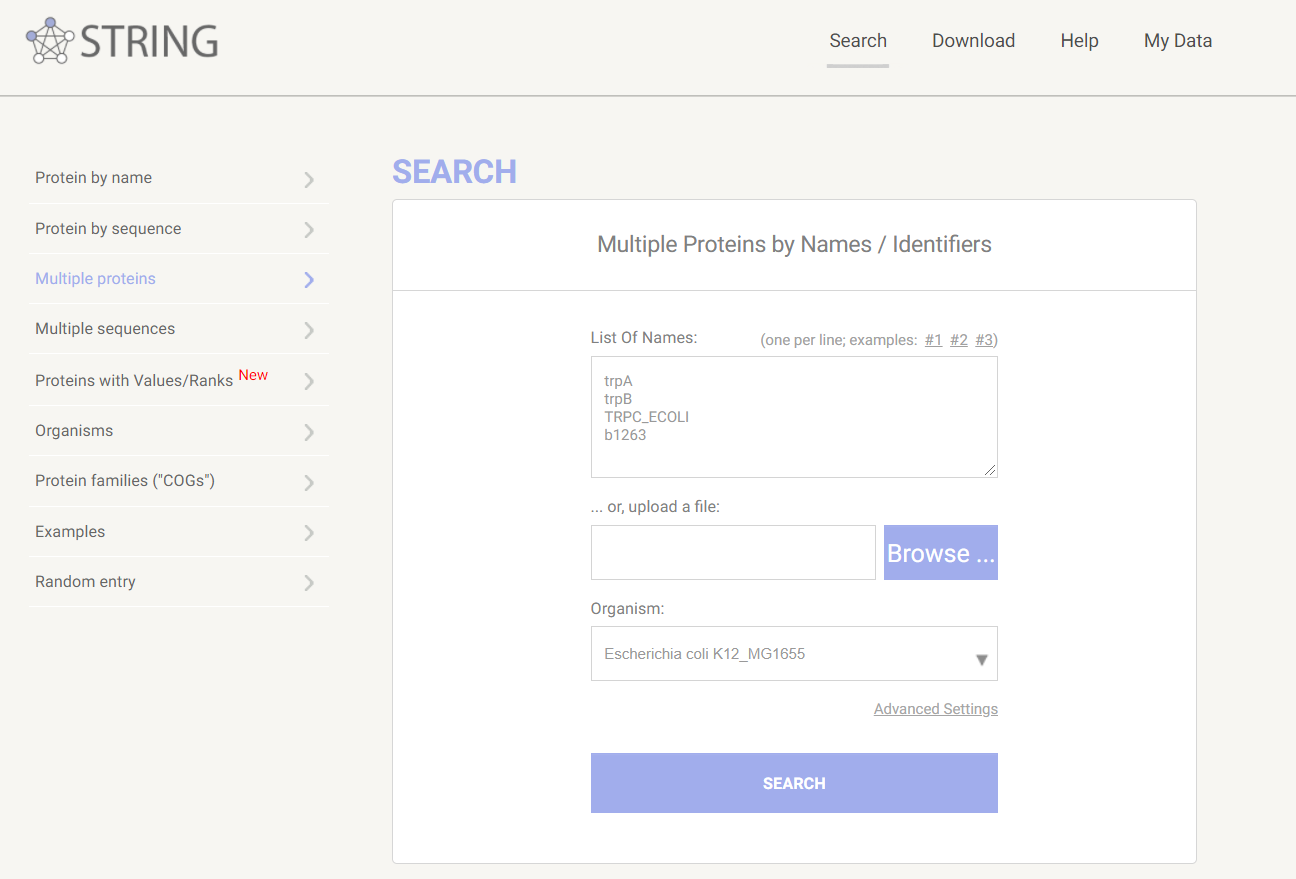
图1

图2
在结果中,点击exports后可以选择下载tsv文件及互作网络图。
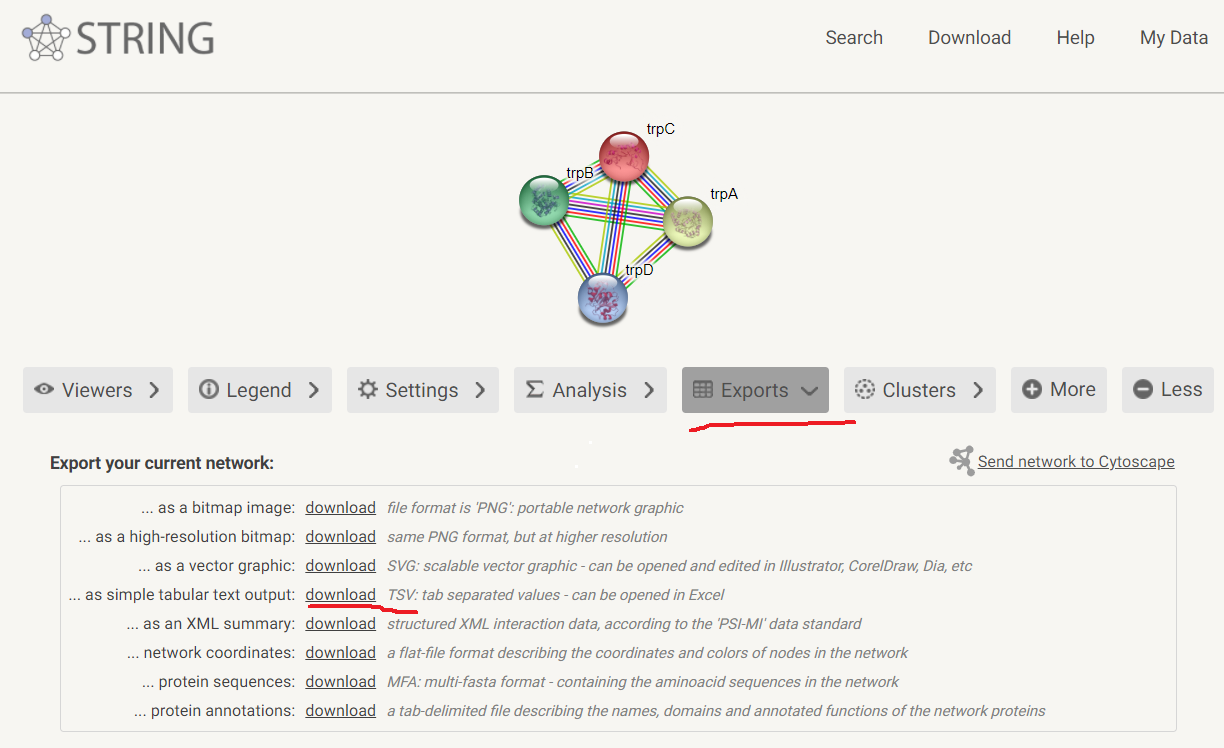
图3
二、cytoscape使用
Cytoscape是一个开放源代码软件平台,用于可视化复杂的网络并将其与任何类型的属性数据集成。在使用之前我们需要下载安装,安装完成后,我们可以导入之前导出的tsv网络文件。
2.1、导入网络文件
打开Cytoscape后,在菜单栏选择file -> import -> network from file,选择string导出的tsv文件即可。
2.2、hub基因查找
hub基因是具有代表性的一些基因,在生物学过程中发挥着比较重要的作用,要分析hub基因,我们需要安装cytohubba app,安装也比较简单,在cytoscape选择app -> app manager,然后查询cytohubba,再点击安装即可。
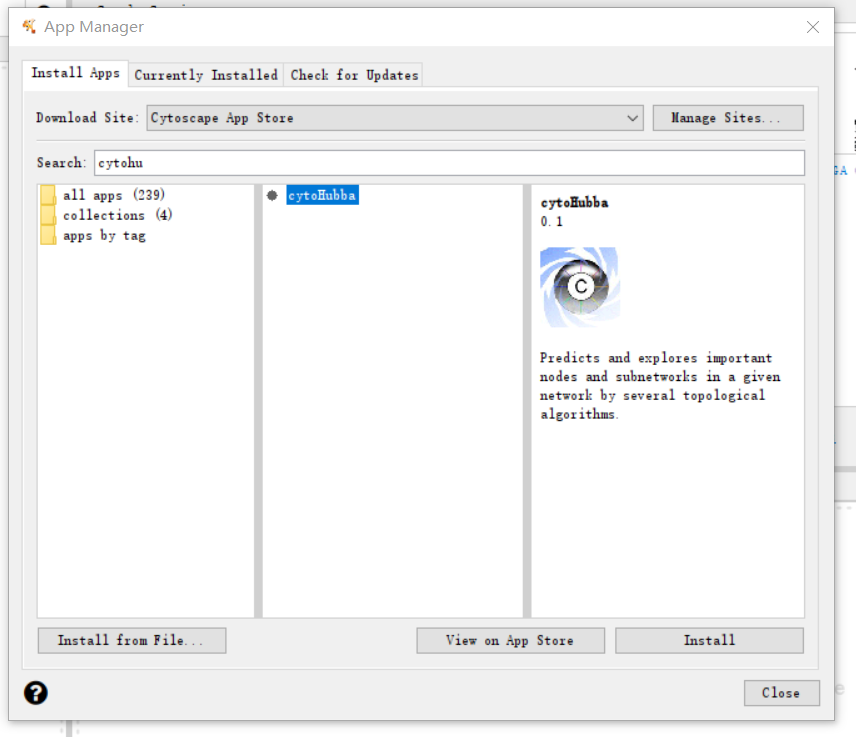
图4
安装完成后,点击左边选项卡选择cytohubba,而后选择对应的网络文件,点击计算,而后再选择显示的top数量,点击submit即可。
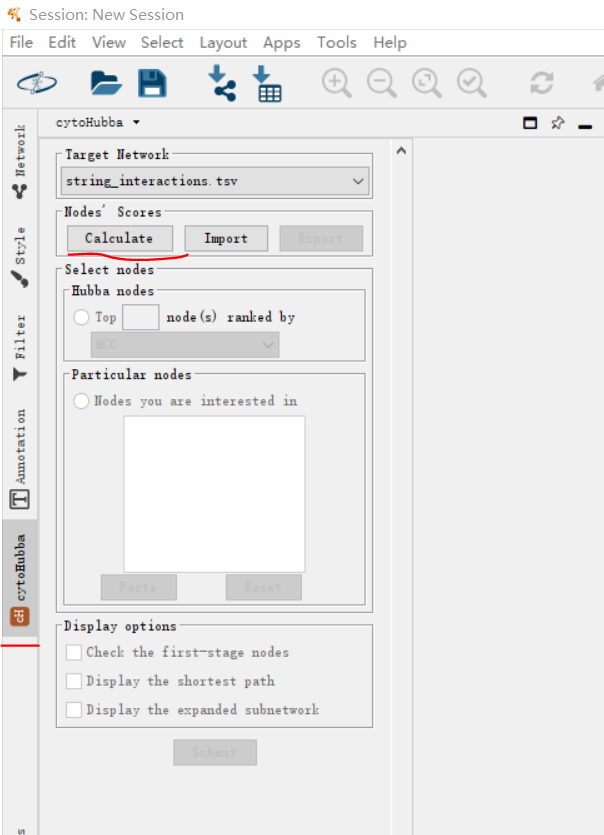
图5
而后即可将rand信息和图片信息导出。
版权声明:本文转载请注明出处!



最新评论:
发表评论
电子邮件地址不会被公开。 必填项已用*标注